
MicroRNA通过调控基因表达和基因组稳定性在生物体内及信号转导途径中发挥着重要作用。最近的数据表明,利用二代测序方法从生物体液中高通量定量miRNAs为发现各种疾病的生物标志物带来了希望,这也将有助于促进癌症的早期诊断。然而,复杂的测序文库构建过程往往会引入偏倚,并可能掩盖miRNA的实际表达水平,从而导致错误性的结论。
近日,北京化工大学许立达教授课题组在Quantitative Biology期刊上发表了题为“The methodological challenge in high-throughput profiling and quantifying microRNAs”的综述文章(点击文末“阅读原文”下载PDF全文)。文章详细讨论了miRNA测序文库构建过程中各步骤的偏倚问题,并提出了多种策略以避免或最小化偏倚,从而生产高质量的数据。此外,文章还对多种市售的miRNA文库制备试剂盒进行了比较。希望本文能为高通量定量分析microRNA的初学者提供帮助。

全文概要
本文首先系统总结了在高通量分析和定量miRNAs过程中所引进的不同来源的偏倚以及优化步骤,并从miRNA文库制备的每个步骤进行了讨论(如图1)。
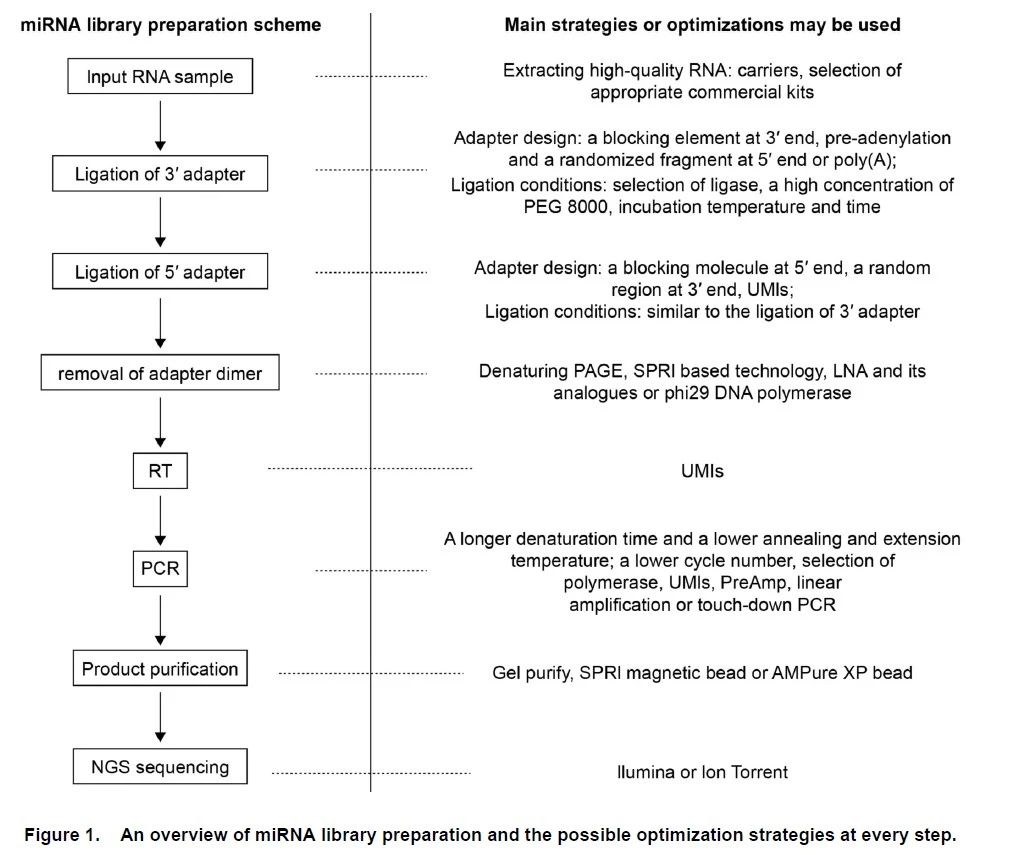
由于cDNA文库制备的众多步骤可能是结果偏倚的来源,因此研究人员倾向使用多种优化方法建立高质量的测序文库,以确保高通量分析和定量microRNA结果的准确性。本文介绍了几种具有代表性的组合优化策略,如TEsR(target enrichment of sRNAs)方法通过引入生物素修饰的RNA探针、优化接头及使用连接酶来确保稀有sRNAs的检测及可用的多种形式的sRNAs,但该方法不适合新型sRNAs的研究。QsRNA-seq(Quantitative small RNA sequencing)使用了优化的SPRI和5‘接头,以及由8个随机核苷酸组成的UMIs,可以非常准确、全面地定量miRNA。Small-seq与传统方法相比,它主要是采用了寡核苷酸修饰的rRNA来避免高丰度5.8SrRNA的干扰;lambda外切酶可减少接头二聚体的形成;5‘接头UMI可去消除PCR扩增和small RNA分子定量的偏倚,但该方法的偏差会来自3’接头末端。AQ-seq(accurate quantification by sequencing)方法通过组合应用4个简并核苷酸和在3‘ 接头和5‘接头连接反应中使用20% PEG,可显著提高miRNA的量,并能检测到低丰度的miRNA。RealSeq-AC方法通过连接一个单端的3’接头及环化miRNA-接头连接产物,从而提高了miRNA定量的准确性。该方法还设计了一种阻断分子阻止了接头的自身环化,从而可从1ng总RNA样品中检测到miRNA。
此外,本文还将市售的一些low-input miRNA文库制备的特点进行了对比,如表1。

希望本文对miRNA文库构建的初学者在实验设计、试剂盒购买及如何避免实验偏倚问题提供帮助。




文章中观点仅代表作者个人观点,不代表本网站的观点和看法。
神州学人杂志及神州学人网原创文章转载说明:如需转载,务必注明出处,违者本网将依法追究。