
在单细胞测序测序技术日趋成熟并被广泛应用于组学研究的今天,由原始测序数据得到的表达谱仍然受到不同处理软件算法的影响而呈现差异。不仅如此,测序平台间较大的技术差异也会进一步扩大算法的影响。为了有效评估不同平台与不同数据处理软件的组合,中国科学院营养与健康所李亦学教授课题组对来自Illumina NovaSeq 6000和华大MGISEQ 2000 两个测序平台的相同文库测序数据分别使用三种常见软件进行了数据处理,随后针对处理得到的表达谱依次从灵敏度、准确性以及下游分析结果三个角度进行评估。结果发现,UMI-tools在不同测序平台数据上均呈现出最好的灵敏度与最多标志基因,另一方面,Cell Ranger在准确性上是三款软件中表现最好的。该评估工作的文章“Comparative analysis of NovaSeq 6000 and MGISEQ 2000 single-cell RNA sequencing data”于近日发表在Quantitative Biology期刊上(点击文末“阅读原文”下载PDF全文)。

全文概要
单细胞RNA测序能够揭示组织中的细胞组成与同种细胞异质性因而被广泛应用于转录组研究。然而受到不同测序平台与处理方法的影响,同一测序样品仍然会得到不同的表达谱,于是评估测序平台与数据处理软件对于理解单细胞表达谱测序结果是十分必要的。现今的评估工作往往局限于测序平台之间或囿于各个数据处理软件之间,尚未有研究能同时兼顾两者。
鉴于此,我们从公共数据库中下载了经由NovaSeq6000与MGISEQ2000两种平台测序的相同样本的数据,并应用三种常见的数据处理软件Drop-seq-tools、UMI-tools、Cell Ranger分别对该数据进行处理从而得到了六种平台-处理流程的组合,最终从灵敏度、准确性、下游分析三个角度计算出评估指标。其中灵敏度被定义为测序深度变化区间内被测到的基因数目(如图1左)、准确性的评估指标我们使用的则是表达缺失的比率(如图1右)。
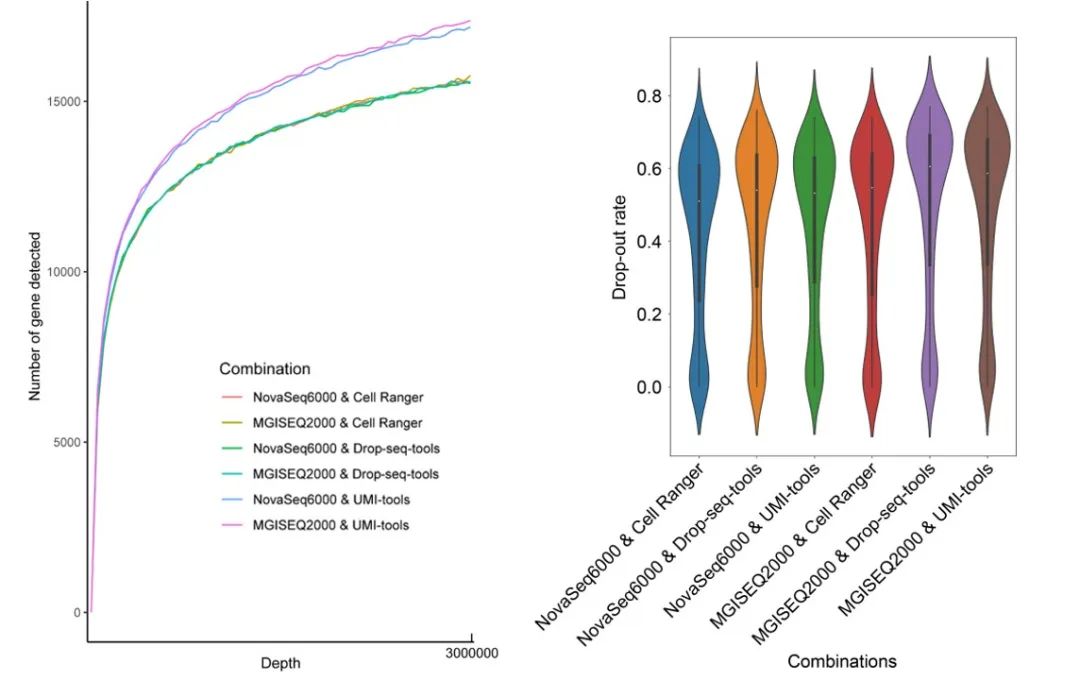
图1,各平台-处理流程组合在灵敏度与准确性上的表现
下游分析被加入了标志基因数、细胞注释数两种指标(如图2),可见不同测序平台数据均能够发现样品中的细胞类型,其中UMI-tools与Cell Ranger在细胞注释上的表现相当。UMI-tools检测到的标志基因最多,这点与灵敏度指标是相互印证的。

图2. 各平台-处理流程组合在下游分析中的表现
综上所述,UMI-tools在不同测序平台数据上均呈现出最好的灵敏度与最多标志基因,另一方面,Cell Ranger在准确性上是三款软件中表现最好的。该研究针对单细胞测序最常见的两类测序平台以及三款处理软件进行了组合评估,从而为不同的研究策略提供测序与分析的指导,同时也为后来的测序评估工作提供了分析框架。


QB期刊介绍
Quantitative Biology (QB)期刊是由清华大学、北京大学、高教出版社联合创办的全英文学术期刊。QB主要刊登生物信息学、计算生物学、系统生物学、理论生物学和合成生物学的最新研究成果和前沿进展,并为生命科学与计算机、数学、物理等交叉研究领域打造一个学术水平高、可读性强、具有全球影响力的交叉学科期刊品牌。
QB期刊目前已被ESCI, Scopus, CSCD等国内外重要数据库收录。Citescore2021=4.6,2023年将获得第一个影响因子(IF)。


文章中观点仅代表作者个人观点,不代表本网站的观点和看法。
神州学人杂志及神州学人网原创文章转载说明:如需转载,务必注明出处,违者本网将依法追究。